东京都立大学的科学家们使用机器学习来自动识别姐妹染色单体凝聚力的缺陷。他们用单个染色染色体的显微图像训练了一个卷积神经网络 (CNN),这些染色体被研究人员识别为具有或不具有内聚缺陷。经过训练,它能够成功地对 73.1% 的新图像进行分类。自动化有望提供更好的统计数据,并更深入地了解导致内聚缺陷的各种障碍。
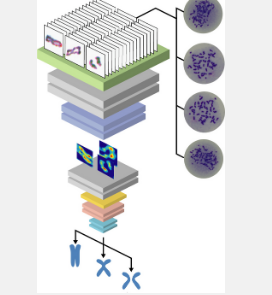
染色体由包含我们基因一部分的长 DNA 分子组成。当细胞分裂时,需要复制染色体,以便两个新细胞都拥有它们发挥功能所需的所有信息。这是通过 DNA 复制实现的,创建两个相同的副本,称为姐妹染色单体,它们通过称为内聚力的环状蛋白质结构结合在一起。在细胞分裂过程中将这些副本保持在一起至关重要。凝聚力问题会导致染色单体分崩离析,从而严重破坏细胞和器官的健康运作。
染色体内聚缺陷的研究主要由研究人员在显微镜下观察染色体进行。使用特殊染色剂,经验丰富的科学家可以判断染色单体是否以正确的方式结合。这种分类对于研究染色体缺陷至关重要,包括正确的凝聚功能。但是,整个过程是手动的。当需要统计有多少染色体处于正确或不正确的状态时,这个过程变得极其低效,需要经验丰富的科学家花费大量的工时。
现在,由东京都立大学的生物学家和机器学习专家组成的跨学科团队由助理教授 Takuya Abe、教授 Kiyoshi Nishikawa、副教授 Kan Okubo 和教授 Kouji Hirota 联合起来,将这一耗时的过程自动化。他们使用支持面部识别和机器视觉的相同技术来分析有和没有内聚缺陷的染色体的显微图像。他们使用卷积神经网络 (CNN),这是一种特别适合图像识别的机器学习算法,并在 600 多张染色体图像上对其进行训练,这些图像已由科学家手动预分类为三组。到该过程结束时,通过算法输入的新图像可以按照与经验丰富的研究人员 73 相同的方式进行分类。1%的准确率。这有可能显着简化和加速染色体实验。
该团队还使用了一种细胞系,该细胞系敲除了一种已知会影响凝聚力的基因,称为CTF18,并使用经过训练的神经网络分析了染色体。该网络发现正常细胞和CTF18敲除细胞之间存在显着差异,表明该网络本身能够发现影响凝聚力的遗传问题。虽然他们的方法目前只能识别三组,但它可以扩展到不同物种的不同模式,从而能够对广泛疾病中的染色体缺陷进行快速分类和前所未有的精确定量。
这项工作得到了 Uehara 纪念基金会、Mochida 医学和药物研究纪念基金会、Kanae 医学促进基金会、Senri 生命科学基金会和 JSPS KAKENHI 资助编号 17K17986、20K06760、22H05072、22K12170、20H04337 的支持, 19KK0210 和 16H01314。
标签:
免责声明:本文由用户上传,如有侵权请联系删除!
